 ConfeitoGUI
相関ネットワーク解析ツール
ConfeitoGUI
相関ネットワーク解析ツール
★ ConfeitoGUIについて
♦ 概要
ConfeitoGUIはスタンドアローンのツールで、
金平糖アルゴリズム(the Confeito algorithm)に基づき、
相関行列データにおいて関連性の高い要素のグループを選び出す。
金平糖アルゴリズムはネットワーク解析手法のひとつで、
局所的なコミュニティを効率的に抽出するアルゴリズムである。
♦ 使用方法
-
Java Runtime Environment (JRE)のインストール(詳しくは取扱説明書を参照)
-
JREのバージョンの確認方法:コマンドプロンプト(端末)で「java -version」
(もしバージョン情報の中に「64-bit」が含まれていなければ、64ビット版のJAVAをインストールする)
-
JRE64ビット版のダウンロード(Windows):こちらから
- 「Windowsオフライン(64ビット)」をクリックして実行
- JRE64ビット版のインストール(Linux):こちらを参照
-
ConfeitoGUIのインストール
- 「ConfeitoGUI_x.x.x.zip」を適当なフォルダに解凍する。
- ツールのダウンロードはこちら
-
ConfeitoGUIの実行
- 上記で解凍されてできたフォルダ内の「launch.bat」をダブルクリックして実行する。
- 解析方法についてはこちら
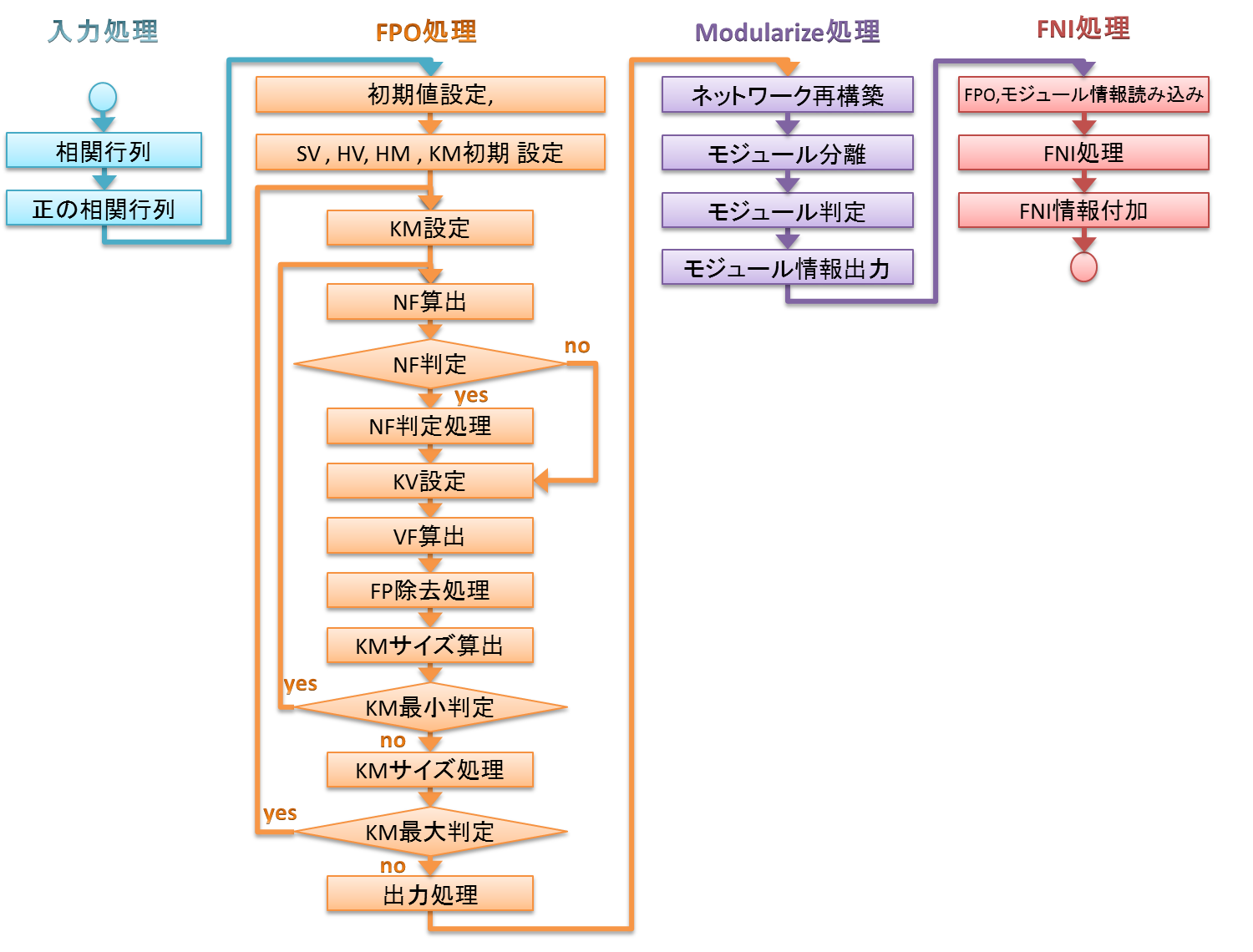
♦ 解析の流れ
- Array Data Converter(オプション): マイクロアレイデータの生データをテキストファイルに変換する
- Correlation Tool: 要素間の相関行列を作成する
- Preprocessing Tool: 要素ごとの相関ファイルのリストを作成する
- Check Tool: 相関ファイルをチェックする
-
FPO (False-Positive-Out) analysis: 互いに高い相関関係を示す要素群(モジュール)を選出する
(ある要素と相関係数の高い要素の中には、どの要素とも相関の高い要素、
すなわち「偽陽性」の要素が存在し、その要素を効率的に省く)
-
FNI (False-Negative-In) analysis(オプション): 個々のモジュールについて、相関係数は低いが特異的に関連している要素を選出する
(あるモジュールとの相関係数は低くFPO解析では検出されないが、特異的に関係している要素、
すなわち「偽陰性」の要素を検出する)
|
 |
♦ 他のツールとの比較
ConfeitoGUIで生成されるモジュールの精度を確かめるために、
NCBIのGene Expression Omnibus (GEO)からマウスのマイクロアレイデータを入手して、
他のコミュニティ抽出ツールとの比較解析を行った。
-
GEOについての基礎知識
- GPL: 遺伝子発現データのプラットフォーム、各社のマイクロアレイや次世代シーケンサー等の種類
- GSE: GPLに含まれる遺伝子発現データの実験群
- GSM: GSEに属する遺伝子発現データ
- GSE内の実験は互いにある程度類似している場合が多い
- すなわち、実験間での相関ネットワーク上で、同じGSEに属する実験(GSM)は
コミュニティ抽出ツールの結果としても同じモジュールに含まれることが期待される
-
使用するデータと相関係数の算出と相関ネットワークの描画
- アフィメトリクス社製のマウスのマイクロアレイGPL1261の
遺伝子発現データ37,013枚を用いた
(データのリストはこちら)
- 遺伝子発現実験間でコサイン相関係数を算出し、相関行列を作成した
- 0.50から0.99まで0.01刻みの相関係数を閾値とした相関ネットワークを描画した
-
比較する他のコミュニティ抽出ツール
- Louvain (Blondel et al., J Stat Mech, 2008):
Pajek上で実行
- Simulating annealing (Newman and Girvan, Phys Rev E, 2004):
R上で実行
- Fast greedy (Clauset et al., Phys Rev E, 2004):
R上で実行
-
比較する方法:モジュール(コミュニティ)と実験群のメンバー構成の類似性を評価する
- メンバー構成の類似性の評価にはF-measureを用いる
-
F-measureは情報科学分野の指標で、
precisionとrecallの調和平均である
-
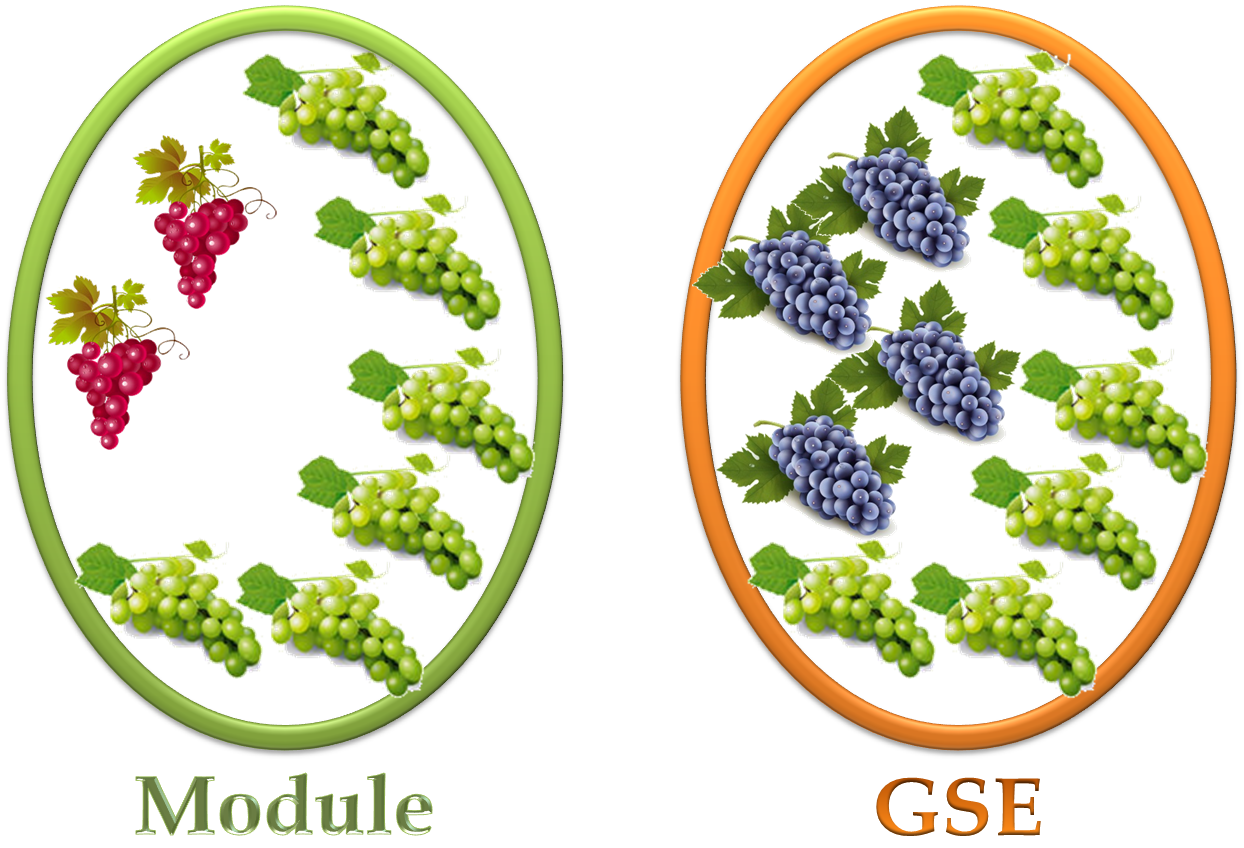
ここでのprecisionは、あるモジュールに含まれる実験
(
  )の中で
特定の実験群(GSE)に属する実験( )の中で
特定の実験群(GSE)に属する実験( )の割合を表す )の割合を表す
-
ここでのrecallは、ある実験群(
  )の中で
特定のモジュールに属する実験( )の中で
特定のモジュールに属する実験( )の割合を表す )の割合を表す
- 下図の例の場合に、precisionは 6 / 8 = 0.75、recallは 6 / 10 = 0.60 となる
-
すなわち、F-measureは( 6 + 6 ) / ( 8 + 10 ) = 0.67となる
- F-meausreが大きいほど、モジュールとGSEの実験群が似ていることになる
- 各閾値の相関ネットワークのすべてのモジュールに対してF-measureを計算した
- 各相関ネットワークごとに、F-measureの平均値を各ツールの代表値とする
- すなわち、この平均値が大きいほど、モジュールの抽出の精度が高いことになる
|
 |
-
比較解析の結果
- 下表はツールごとのF-measureの平均値を表している
| Tool | F-measure (average ± SD) |
| ConfeitoGUI | 0.61 ± 0.34 |
| Louvain | 0.47 ± 0.40 |
| Simulating annealing | 0.40 ± 0.36 |
| Fast greedy | 0.37 ± 0.37 |
- ConfeitoGUIが最も高い平均F-measure値を示しており、モジュールの精度が高いことを表している
♦ 文献
-
Ogata Y et al., ConfeitoGUI: a toolkit for community detection from a correlation network in molecular biology,
with a function to adjust community size. (submitting)
-

 Sakurai N et al., KaPPA-View4: a metabolic pathway database for representation and analysis of correlation
networks of gene co-expression and metabolite co-accumulation and omics data.
Nucleic Acids Res, 39: D677-D684, 2011.
Sakurai N et al., KaPPA-View4: a metabolic pathway database for representation and analysis of correlation
networks of gene co-expression and metabolite co-accumulation and omics data.
Nucleic Acids Res, 39: D677-D684, 2011.
-

 Ogata Y et al., CoP: a database for characterizing co-expressed gene modules
with biological information in plants. Bioinformatics, 26(9): 1267-1268, 2010.
Ogata Y et al., CoP: a database for characterizing co-expressed gene modules
with biological information in plants. Bioinformatics, 26(9): 1267-1268, 2010.
-

 Ogata Y et al., The prediction of local modular structures in a co-expression network
based on gene expression datasets. Genome Inform, 23(1): 117-127, 2009.
Ogata Y et al., The prediction of local modular structures in a co-expression network
based on gene expression datasets. Genome Inform, 23(1): 117-127, 2009.
♦ 拡張版
本ツールはアカデミックフリー版であり、拡張版(ConfeitoGUIplus)については、かずさDNA研究所に問い合わせてください。
 |
|
The KAGIANA Project |
|
(since 2006) |
Copyright (C) 2015 All rights reserved Osaka Prefecture University
& Kazusa DNA Research Institute
 ConfeitoGUI
相関ネットワーク解析ツール
ConfeitoGUI
相関ネットワーク解析ツール
 ConfeitoGUI
相関ネットワーク解析ツール
ConfeitoGUI
相関ネットワーク解析ツール

