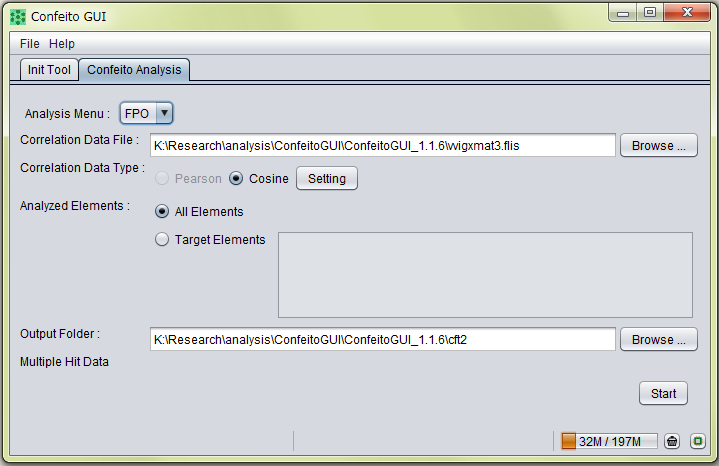
 ConfeitoGUI
相関ネットワーク解析ツール
ConfeitoGUI
相関ネットワーク解析ツール
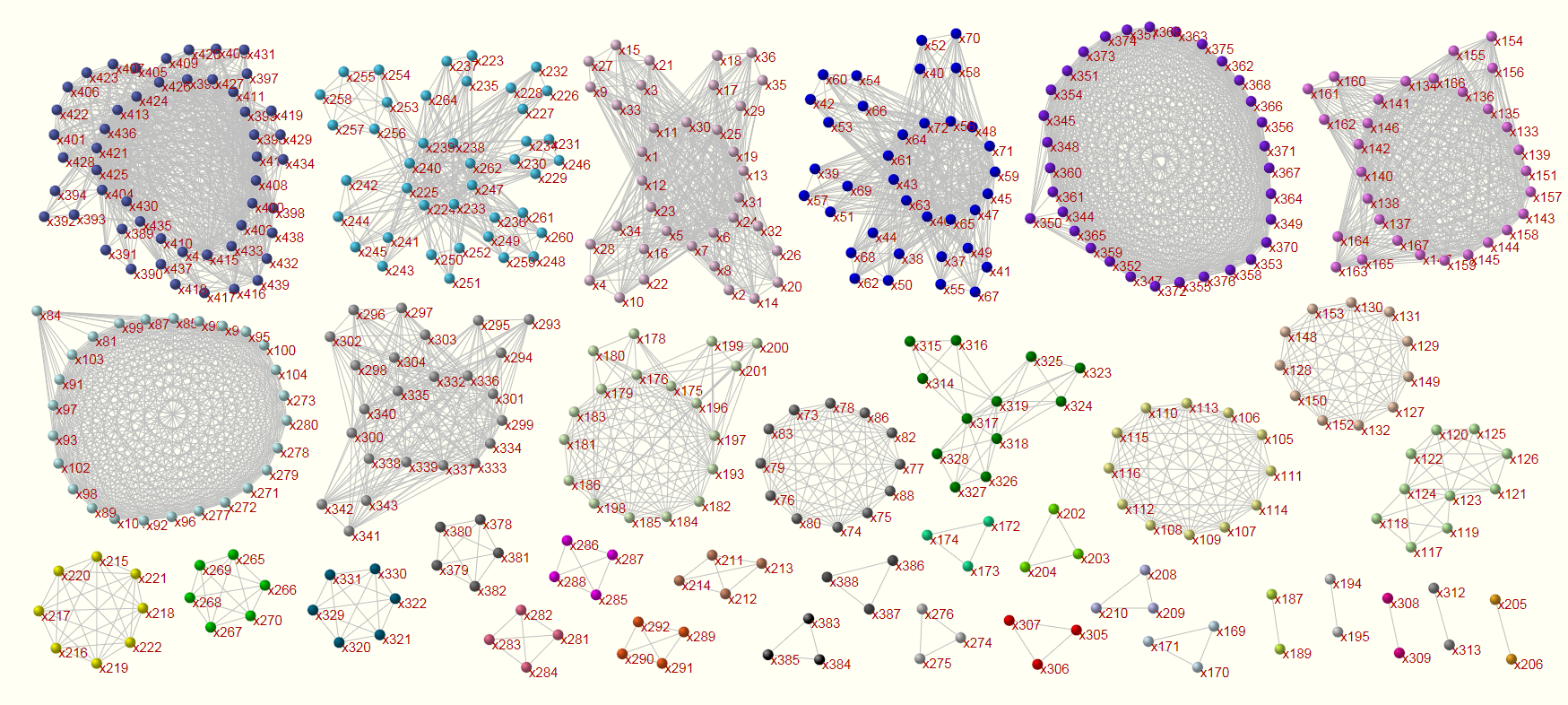
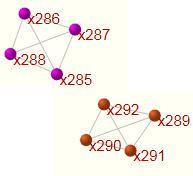
 ConfeitoGUI
相関ネットワーク解析ツール
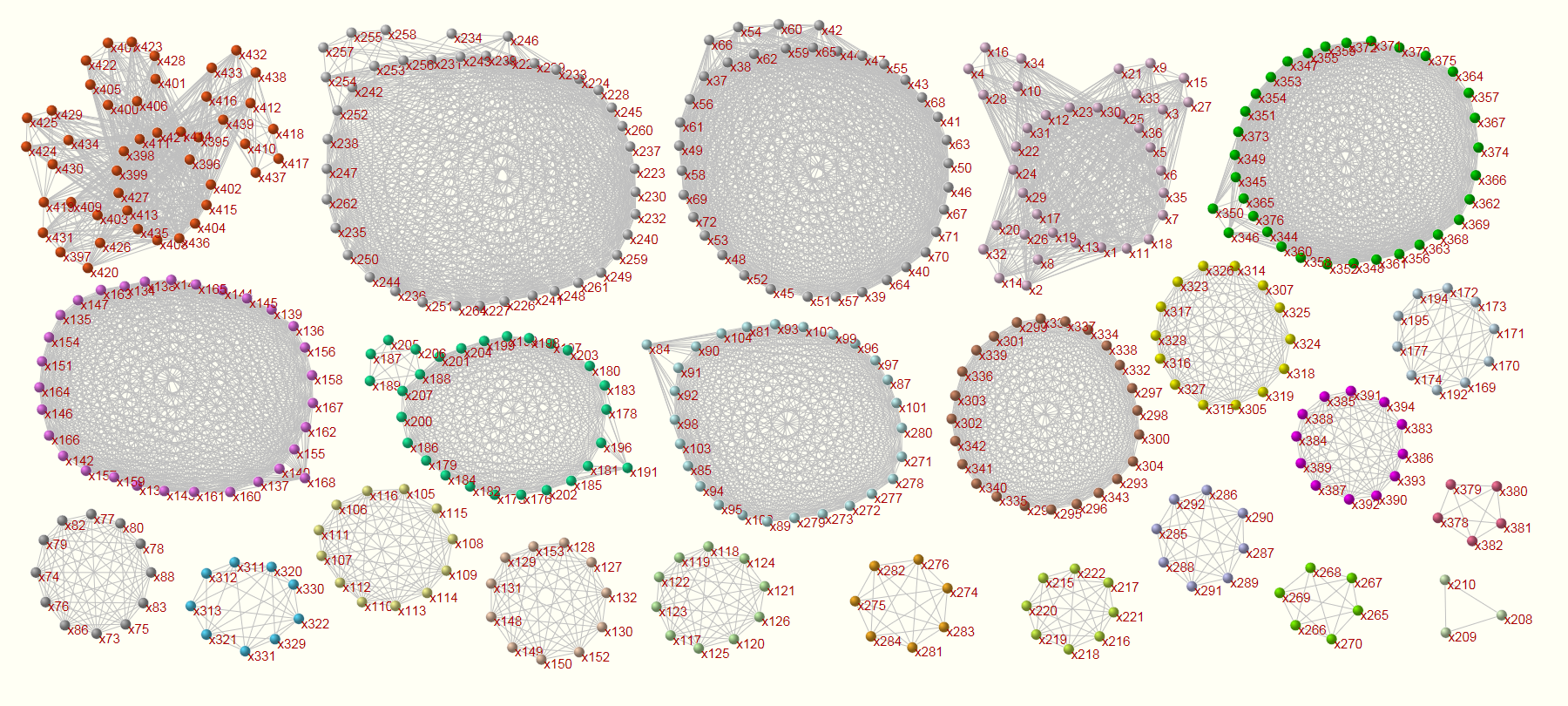
ConfeitoGUI
相関ネットワーク解析ツール
|
|
|
|
|
|
|
|
|
|
|
|
|
|
| The KAGIANA Project | (since 2006) |